2022/4に、ImageJでRadiomic特徴が計算できる「RadiomicsJ」というJavaのライブラリを公開しました。
RadiomicsJはImageJやFijiのプラグインインターフェースがインプリメントされており、ImageJ/Fijiプラグインとして動作します。 今回はこのRadiomicsJを使ったRadiomicsをご紹介いたします。
Radiomicsとは
レディオミクスは、「高信号/低信号」「不明瞭」「辺縁に造影効果」「均一/不均一」などの医用画像から得られる特徴が、分子生物学的特徴のパターンを捉えることができるという仮説に基づき、腫瘍遺伝子型や予後などの予測やイメージングバイオマーカの発見などを目的として行われる研究手法です。
類似した研究領域として、従来から、コンピュータ支援診断(computer-aided diagnosis, CAD)がありますが、レディオミクスはCADの中の一つというよりも、独立した研究として説明されるように思います。筆者の個人的な、CADとレディオミクスとの違いについての認識は次の通りです。CADは、良悪性鑑別予測、異常検知、予後予測などを主な目的として、医師の意思決定支援のために研究されてきました。これに対してレディオミクスは、目的変数をこれらに限定せず、分子生物学的特徴という概念に置き換えた上で、マクロな画像の世界と細胞やDNAなどのミクロな世界の関係の探索を目的として研究されています。
レディオミクスはだれでも試行できます。例えば、TCIA(The Cancer Imaging data Archive)などからオープンデータセットを探して、脳MRI画像を使って画像特徴を計算し、この計算結果から脳腫瘍のWHOグレードを予測したり、腫瘍遺伝子型の異常の有無を予測したりなどです。
PubMedなどで検索をすれば、他にもさまざまな応用例がヒットします。
しかし、いざやってみようとなると、なにから行えばよいかわからなくなることがあります。
本ブログでは、「胸部レントゲン画像の肺野・縦隔から60歳以上かどうかを予測する」ことに挑戦してみましたので、手順を公開いたします。
環境
Windows(Mac、Linuxでも動作するはずです)
必要なもの
- ImageJ
- ImageJサイト(https://imagej.nih.gov/ij/download.html)の説明に従ってダウンロードします。
- RadiomicsJ
- こちらからダウンロードします。
- https://drive.google.com/file/d/148ToZ1knJOpi4pzIwSf8g23kn-d006tR/view?usp=sharing
- ダウンロードしてzip解凍後します。
図 RadiomicsJを解凍して内容を展開した様子
- 「RadiomicsLib」フォルダと「My_RadiomicsJPlugin.class」ファイルを、インストール済みのImageJのpluginsフォルダにコピーします。
図 ImageJのpluginsフォルダへコピー(これでインストール完了)
- JDK 1.8
- OpenJDK1.8の最新バージョンをインストールして、環境変数にJAVA_HOMEパスを追加してください。 (JDK 1.8以外ではエラーが起こる可能性があります。)
- 参考:JDKのインストール方法(Windows)https://www.javadrive.jp/start/install/index4.html
- データセット
- こちらからダウンロードします。
- https://drive.google.com/file/d/1Tet5rSuD1Zw1tbyIicGGU3dyYIO5OIht/view?usp=sharing
- 画像データ、Ageラベルデータ、解析処理のsettingsファイルが含まれています。
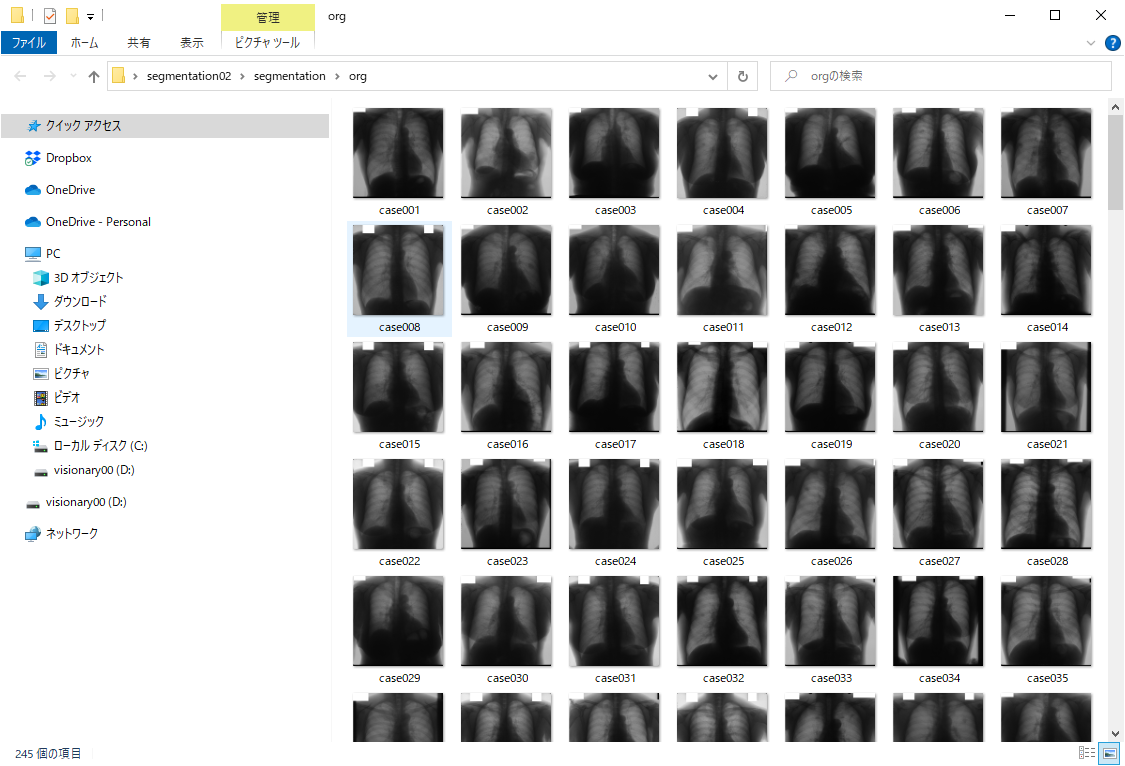
- データセットの元は、JSRTから教育研究用に公開されているSegmentation02データセットおよびAgeデータセットです。 ダウンロード用のデータセットは、元のデータから欠損ラベル除外、ファイル名統一を行ったデータになっています。
- Weka
- こちら(https://waikato.github.io/weka-wiki/downloading_weka/)からOSに合わせてダウンロードします。
レディオミクスのプロセスの全体像
レディオミクスのプロセスは、大きく二つのステップで構成されます。
- 画像特徴を計算する
- 機械学習で予測モデル作成する
①は、解析したい画像上の関心領域を決めて、画像特徴を計算します。
図 胸部レントゲン画像(case001)
今回の胸部レントゲンの例では、解析したい画像上の関心領域はマスク画像(ラベル画像とも呼びます)で指定されます。今回の例では、肺野領域:255、心臓:85、肺野外:170、体外:0の画素値になっています。肺野は、マスク画像上で255の値の白い領域です。この肺野から画像の特徴を計算したいときは、この領域を計算するように設定すればよいということになります。心臓の縦隔領域を解析したい場合は、85の領域(濃いグレーの部分)を指定すればいいということです。
②の機械学習は、一般的な機械学習の手順で、説明変数と目的変数を設定して、予測モデルを作成・テストします。
レディオミクスで一番大変なのは、このようなデータセットの準備ですが、ImageJを使って比較的簡便に作成できます。
ここで、マスク画像の作成方法を紹介します。(この操作は以降の機械学習モデル作成には不要です)
作成時のポイントは、ROIマネージャツールを使うことです。
ROIマネージャはAnalyze>Tools>ROI Managerから起動します。ROIマネージャにROIを追加するには、好きなROIツールで画像にROIを描いた後、「Add」します。
(描かれたROIはROIマネージャにAddされている)
ROIを更新したとき(形や位置を変えたとき)は、「Update」ボタンを必ず押すようにします。
ROIが設定出来たら、ROI以外の領域を0、ROIの内側を任意の値にします。
RadiomicsJは8-bitグレースケールのマスク画像(Tifがデフォルト)に対応していますので、ROIの内側の値は、0~255である必要があります。
例えば、肺の領域を255、そのほかを0にしたい場合は、ImageJメニューのEdit>Clear OutsideでROI以外の領域を0にできます。
Clear Outsideをしてから、Process>Math>Set...で、値を255にして実行します。
完成したら、解析画像とはフォルダを分けて、Tif形式で保存しておきます。保存するときの画像ファイル名は、解析画像と同じ画像名などにしておくとわかりやすいです。RadiomicsJはTif形式が標準画像フォーマットです(ImageJで読み込めるファイルならばOKです)。保存の際、元の解析画像を上書きしないように注意します。 ROIデータの結合や分離もROI Manager上で行うことができます。ROIデータは保存しておくことができるので、やり直しも可能です。
Radiomic特徴を計算する
RadiomicsJによるRadiomic特徴の計算には、3つのデータが必要です。
- 画像データ
- マスクデータ
- セッティングファイル
RadiomicsJはグラフィカルユーザーインターフェースからの操作や、コマンドプロンプトから一括計算処理することもできます。
セッティングファイルはコマンドプロンプトによる操作の時に必要になります。順に解説します。
ImageJのプラグインとして利用する方法(一例ずつ処理)
画像を一枚一枚確かめながら解析を行いたい場合は、解析画像とマスク画像をIJで表示した状態で、ImageJのプラグインを起動し、画像を設定して、計算を実行します。
図 Plugins>My_RadiomicsJPlugin (解析画像とマスク画像はプラグインを起動する前にIJに表示しておく)
追加で解析をしていく場合も同様の操作を繰り返します。 このとき、ResultTableを閉じずにそのままにしておくと、行が追加されていきます。
結果を保存したいときは、このResult Tableの機能(File>Save)を使って保存できます。
しかし、一般に、大量のデータを自動的に繰り返し処理したいことの方が多いと思います。そのような場合は、次に説明するコマンドプロンプトからの操作が可能です。
コマンドプロンプトから利用する方法(自動で全処理)
コマンドプロンプトから利用する場合、データセットをフォルダに綺麗に分けておく必要があります。綺麗にというのは、画像とマスクがセットになるように、症例ごとに分けておくという意味です。この際、画像とマスクのファイル枚数は同じになる必要があります。
今回の画像データセットはあらかじめorgとlabelにフォルダが分かれており、対応する画像ファイルがそれぞれに準備されています。ファイル数も同じになっています。
(マトリクスサイズが異なる場合は先に整えておくか、症例ごとにフォルダを分けておく必要があります。また、CTやMRIのようにシリーズが複数の画像で構成される場合もフォルダで分けておく必要があります。 )
あとは、計算に必要なセッティングをします。
計算の実行には、必要に応じて、セッティング情報(.propertiesファイル)を指定できます。
RadiomicsJは3Dがデフォルトです。そのため、今回の胸部レントゲンのような2D画像を解析するときは2Dで解析するように設定します。
CTやMRIのシリーズ画像を3Dで処理する場合は、セッティングファイルは省略可能です。
今回のデータセットはすべての画像の縦横のマトリクスサイズが同じですので、一連として一括処理します。
セッティングファイルの内容
セッティングファイルには、RadiomicsJで計算可能な特徴ファミリーの計算設定をセットします。 よく使うのは、マスクのラベル値の指定(INT_label)、2D解析とするかどうか(BOOL_force2D)、2Dの画像特徴を計算するか(BOOL_enableShape2D)、デフォルトでない特徴を有効にするか(BOOL_activate_no_default_features)です。
有効無効の設定は、1(True)、0(False)で指定します。
今回は、2Dでの処理に合わせるために、BOOL_force2D=1, BOOL_enableShape2D=1としています。
セッティングファイル内で、「!」「#」が行の先頭についている行はコメントアウト(その行を無視する)を意味しています。
他の項目は、特徴計算の専門家が設定するような項目なので、最初は気にしなくてもよいです(例えば、32-bitのPET画像を解析するときはBinWidthで実行したほうがよいなど、モダリティによっても考慮すべきことがあります。詳しくはIBSIリファレンスを参照ください)。
特徴計算を実行していきます。
コマンドプロンプトを開き、カレントディレクトリをRadiomicsJLibフォルダにします。
下のようにコマンドを入力し、Enterで実行します。
>cd "RadiomicsJLibフォルダまでのパス"
次に、処理を実行するためのコマンドを入力し、Enterで実行します。
>java -jar RadiomicsJ.jar -i "orgフォルダまでの絶対パス" -m "labelフォルダまでの絶対パス" -s "settings.propertiesまでの絶対パス" -d
- オプションの説明
- -i:解析画像フォルダパス
- -m:マスク画像フォルダパス
- -s:セッティングファイルパス
- -o:解析結果CSV保存先(.csvまで必要)
- -d:処理経過の表示
パスというのは、PC内のフォルダやファイルの場所のことです。
解析結果CSVはデフォルトでは自動で保存されません。自動的に保存したい場合は、-oオプションでcsvファイルの保存場所を指定しておきます。
処理が始まると、計算中のプロセスが表示されます。
245件の処理が終わるまで継続されるので、小一時間かかります。
処理が終わると、Rediomics Features結果テーブルが表示されます。この結果テーブルのFileメニューから結果をCSV保存します。
図 処理結果例(今回はDICOMデータではないので、基本情報はNaNになっている)
これで、画像特徴の計算は完了です。
機械学習モデルを作成する
先に、学習データを完成させます。
今回は年齢を推定するのですが、データも少ないので、分類モデルとして扱う例で示します。60歳以上かどうかを予測するためのモデルを作成してみます。
Radiomic特徴の計算結果CSVをエクセルなどで開き、データセットに含まれているage.csvファイル内にある目的変数「elder_class」を、計算結果CSVの一番左のカラムに追加します。 Ageの情報はファイル名順に並んでおり、解析結果もファイル名順に並んでいますので、そのまま列を挿入して追加します。
次に、不要なカラムを削除しておきます。RadiomicsJはデータの基本的な情報なども取得してテーブルにまとめてくれますが、これらは学習には不要なデータですので、先に削除しておきます。
ここでは、OperationalInfo_ではじまるカラム、Diagnostics_ではじまるカラムはすべて削除します。
この他にも、①相関の強い、または完全相関する変数は削除、②すべてのデータで同じ数になっている(例えばすべて0であるなど)変数は削除、などしてデータを調整しますが、ここでは、この手順を省略します。
ここまでできたら、CSVを別名で保存しておきます(今回はRadiomics Features lung ML-2.csvとして保存しました)。
これで、練習用の学習データの準備が整いました。
Wekaを起動しましょう。
「Explorer」を起動します。Preprocessタブ内の「Open file...」から学習データCSVを読み込みます。 CSVを読み込むために、「Files of Type:」で読み込むファイルの形式をCSVに変更します。
データのサマリーは「Visualize」タブから確認できます。
次に、「Preprocess」タブで、前処理を実行していきます。今回は、定数のみを含むの説明変数の除外のために「RemoveUseless」とデータの標準化「Standardization」を適用してみます。(外れ値の除外も可能ですが、説明が煩雑になるため省略します。方法はこちら:https://youtu.be/WrjpO7CmUoQ)
「RemoveUseless」は、PreprocessタブのFilterボタンから、unsupervised>attributes>RemoveUselessを選択し、右側のApplyボタンを押して実行します。
この操作で、すべて「0」などの値となっていた変数がドロップされます。
次に、同様の操作で、標準化を実施します。unsupervised>attributes>Standardizeで選択できます。同様にApplyして実行します。文字列のカテゴリ変数でない数値の変数はすべて標準化されます。
最後に、学習方法を設定して学習を実行します。 利用する分類器は今回はランダムフォレストにしました。
学習の設定
- Classifier: RandomForest
- Cross-validation: 10 folds-CV
- Target class: elder_class (間違えやすいので注意)
- Startで実行
分類精度は学習終了時に表示されます。
AUCは0.679でした。
Startボタン下にある「Result list」に解析結果が保存されます。このリストを右クリックして、「Visualize thresholdCurve」(TRUEの方)を実行すると、ROC曲線を確認できます。
今回は、分類精度が低くなってしまったのですが、これがAUC 0.9などの予測モデルであれば、結構いい感じの予測モデルになりそうということがわかります。
訓練した予測モデルは、modelファイルとして保存(Result listを右クリックしてSave model)しておき、Weka APIを通じてアプリケーションから利用できます。
Knowledge Flowを使う
Wekaには、複雑な機械学習プロセスを可視化しながら実行できる機能があります。
「Knowledge Flow」です。
この機能は、Explorerでできることをアイコンを使ってパイプラインを描くことができます。Explorerでは、途中でどこまでやったかわからなくなってしまったりすることもあるので、直感的にわかりやすいインターフェースは重宝されます。
今回の解析に特徴選択というプロセスを追加した例を示します。
(Wekaはいろいろな特徴選択が可能です:https://youtu.be/Pf9yKjSiVnw)
- 利用したい処理をツリーから選び、キャンバスに追加していきます。
- 右クリックで処理の流れに使うデータセットを選び、繋げていきます。
- 処理の結果や、マップは、Visualize機能を使います。
- 各アイコンには詳細設定を追加できます。
- フローが完成したら、実行ボタンで実行できます。
縦隔から60歳以上かどうかを予測する
これまでの例では、肺野から取得されたラジオミクス特徴で予測モデルをテストしていました。同じ手順で縦隔(主に心臓)のラジオミクス特徴から60歳以上かどうかを予測することもできます。ラジオミクス計算時に、settings.propertiesの「INT_label」を85にして計算するだけです。
また、AgeラベルCSVには10歳刻みのカテゴリを予測するためのラベルも用意しています。マルチクラス分類は、筆者が試した限りでは難しそうでしたが、ご興味のある方は、ラベルを入れ替えて試してみることができます。
他の秀逸なレディオミクスのツール
RadiomicsJ以外にもGUIから操作できるツールがあります。
- LifeX
- PyRadiomics + 3D Slicer
- etc...
レディオミクスの詳しい話について
IBSIリファレンス(https://theibsi.github.io/documentation/)に詳しい手順が解説されていますので、専門家になりたい方は一読をおすすめします。
おわりに
自分の研究なんて無効な研究だと思っても、無効の組み合わせが有効なものへと形を変えることがあります。
医用画像を使った研究でなんかやろうかなと思っている方は、レディオミクスなんていかがでしょうか。
- 医師
- 治療や診断の高品質化に直結するような研究のために。 研究の成果を積み上げるために。
- コメディカル
- 専門の領域で、治療や診断とは区別したうえで、生物学的な探索や診断や治療を支援するための研究に。 (医師でなくても、オープンデータを使ってさまざまな検討が可能な時代になりました。)
- 最近は、予測モデルをクラウドから実行できるようなサービスもありますので、自分で作った予測モデルを市場に出すことも夢ではなくなりました。
- 情報系エンジニア
- 医療の知識は医療系の人たちから吸収すれば、あとは得意な情報処理。 医療応用のための基礎技術特許などを通じた社会貢献のために。
- バイオ系研究者
- 人と自然との関連の探求に。
RadiomicsJの改良(特徴の追加など)、バグ報告など、ご意見・コメントお待ちしております。
Visionary Imaging Services, Inc.
Tatsuaki Kobayashi
References
- 元データセット:http://imgcom.jsrt.or.jp/minijsrtdb/
- IBSIリファレンス:https://theibsi.github.io/documentation/
- RadiomicsJ(論文引用をしていただけますと幸いです):https://pubmed.ncbi.nlm.nih.gov/35792994/

































0 件のコメント:
コメントを投稿