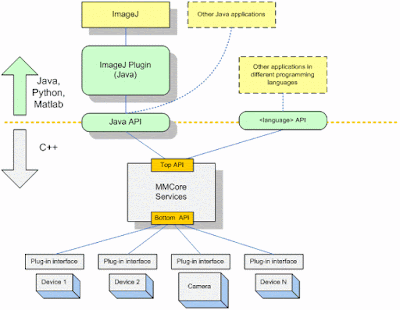
今回は、ImageJのプラグインでも紹介されている特徴点追跡アルゴリズムについて説明します。
特徴点抽出の概要
複数の画像間の対応点を抽出するために局所特徴量を計算する方法には幾つかの手法があり、改良版や高速計算のアルゴリズムなどがいくつかの論文で説明されています。
まず最初に、Fijiによる2値画像間に対応する特徴点の自動抽出結果を示します。
(Plugins>Feature Extraction>Extract SIFT correspondences)
テスト画像(multiple sclerosis):MAGNIMS consensus guidelines on the use of MRI in multiple sclerosis—establishing disease prognosis and monitoring patients, Fig.1(a〜d)
Baseline
Follow up (3 years later)
(計算設定)
特徴点抽出結果(Baseline)
特徴点抽出結果(Follow up (3year))
ここから、この結果がどのように計算されたかを解説していきます。
画像間の相互の特徴点を抽出し、それぞれの点の追跡を行う際のもっとも単純なケースは、対象となる画像同士について、画像サイズが同じで、平行なズレの場合です。その場合は、差分をとってその勾配の方向に移動すればいいということになりますが、画像間で画像サイズが異なる場合や、回転やピクセル値が複雑に変化する場合には、特別なアルゴリズムを適用しなければうまく特徴点を追跡できません。
D.Loweらは、下記に示す、画像特徴量セットを計算するためのステップを説明しています。(ところどころ、オーバーオール?スケール?モデル?という疑問はあると思いますが、詳しくは、参考文献1をご参照ください。)
1.スケールスペース極地検出
これは、オーバーオールのスケールと画像の位置を探索計算するための第一段階です。
スケールや方向に対して不変の関心位置を同定するために、ガウシアン関数の変化を利用して実装しています。
2.キーポイントの位置決め
それぞれの候補点において、モデルが位置とスケールを決定するためにフィッティングされます。キーポイントはそれらの安定性の計測をもとに選択されます。
3.方向の割り当て
1つもしくはそれ以上の方向は、局所画像のグラディエントをベースにそれぞれのキーポイント位置に割り当てられます。すべての先行演算はそれぞれの特徴点ごとに割り当てられた方向、スケール、位置に関連して変換され、画像上で計算されます。
その結果、これらの変換に対して不変性が与えられます。
4.キーポイント記述子
局所画像のグラディエントは、それぞれのキーポイント周辺の領域に選択されたスケールで計測され、局所の形状の歪みや、輝度の変化の有意レベルを許容する代表値に変換されます。
D.Loweらは、局所特徴量に関連したスケール不変軸の中へ画像データを変換するこのアプローチを、”Scale Invarian Feature Transform(SIFT)”と名付けています。
SIFTアルゴリズム
SIFTアルゴリズムについては、藤吉らの研究報告によって日本語で詳細に紹介されています。本稿では、スケールに不変な画像処理を行い、その画像上から特徴点の位置推定、方向および強度を求める計算のロジックを解説します。
まずはじめに、対象画像(群)に対して、Difference of Gaussian処理を行います。これは、単に、σ値を変換して、ガウシアンフィルタを掛けた画像同士を順次サブトラクションしていく処理です。ガウシアンフィルタのσ(標準偏差)が大きくなると、計算量の負担が大きくなるため、σが大きくなるに従って、画像サイズを小さくしていく方法がD.Loweらの高速計算のためのロジックです。
入力画像をI(u,v)、ガウシアン関数をG(x,y,σ)、平滑化画像L(u,v,σ)、平滑画像間の差分(DoG画像)とすると、DoGの結果画像D(u,v,σ)は、下記の式で表されます。
L(u,v,σ)=G(x,y,σ)×I(u,v)
D(x,y,σ)={ G(x,y,kσ)-G(x,y,σ) }×I(u,v)
ここで、σが大きくなると、ガウシアンフィルタのウィンドウサイズが大きくなり、これに伴い、処理ができない端の領域の拡大と計算コストが増加するという問題を解消するために、画像をダウンサイズしてσを落としていく計算を繰り返します。
σ(初期値)を増加させながら、複数の平滑化画像を作成し、例えば、2σ、すなわち、[L1(2σ)]になったら、画像サイズを1/2にダウンサンプリングします。この画像を改めて[L2(σ)]として、オリジナルの画像の時と同様に2σまで計算を繰り返します。
このとき、この計算を適用できるのは、下記の式が成り立つためです。
L1(2σ) ≒L2(σ)
次の図に平滑化処理の例を示します。
最適な平滑化の処理の回数やσの値についても実験から算出されています。
まず、オリジナルサイズの画像の平滑化(オリジナルサイズなので1オクターブと称し、ダウンサイズするたびに2オクターブ、3オクターブとつけていく)の回数を分割数s、σの増加率をkとした場合、kは分割数乗法逆元で増加します。
後述する通り、特徴点となる画像の極致探索には3画像を1セットで処理(26近傍の極致探索を行う)するため、s枚の極致検出の対象となる画像を得るには、s+2枚のDoG画像、必然的にs+3枚の平滑化画像が必要になります。次の図に、藤吉らが提示した説明図を引用します。
(参考文献3より引用)
(参考文献3より引用)
参考文献1によれば、分割数r=3、初期値σ=1.6のときに最適なキーポイントを得ることができるとされています。
参考文献3の図5より、極値には注目点(黒色のピクセル)とその26近傍(灰色)を比較して注目点が極値だった場合は、キーポイントの候補点となります。このとき、極致はσの小さい画像から順番に計算していきます。キーポイント候補点をより厳密に絞り込む方法として、ヘッセ行列を用いた絞り込みが用いられているようです。
次回は続けて、特徴点計算の際のキーポイントの絞り込みアルゴリズムや方向の算出方法を続けて解説します!
参考文献
- David G. Lowe:Distinctive Image Features from Scale-Invariant Keypoints.
- 藤吉研究室中部大学工学部情報工学科.画像局所特徴量と特定物体認識- SIFTと最近のアプローチ -(http://www.vision.cs.chubu.ac.jp/cvtutorial/PDF/02SIFTandMore.pdf)
- 藤吉 弘亘.中部大学工学部情報工学科:Gradient ベースの特徴抽出-SIFT と HOG-(http://www.vision.cs.chubu.ac.jp/SIFT/PDF/sift_tutorial.pdf)
参考記事:「山本修司:ImageJで学ぶ実践医用・バイオ画像処理.INNERVISION(25・4) 2010, p80-81」